dMR预处理全流程
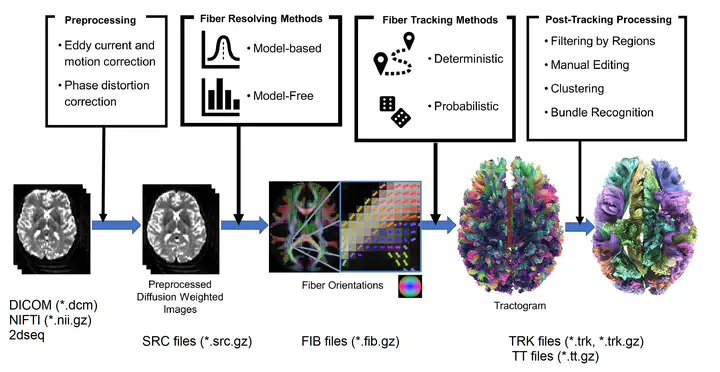
dMR预处理全流程
感谢博士生丛士清参与编撰本文
前提:
输入数据须整理为BIDS格式
示例:BIDS根目录为/bids_root
1. BIDS-dMRIrecon安装
需要安装docker,可以参考https://github.com/chenfei-ye/BIDS-dMRIrecon/blob/main/resources/docker_install.md
方式一:拉取镜像 docker pull mindsgo-sz-docker.pkg.coding.net/neuroimage_analysis/base/bids-dmrirecon:latest docker tag mindsgo-sz-docker.pkg.coding.net/neuroimage_analysis/base/bids-dmrirecon:latest bids-dmrirecon:latest
方式二:镜像创建
# git clone下载代码仓库
cd BIDS-dmrirecon
docker build -t bids-dmrirecon:latest .
2. sMRIPrep批处理T1影像
docker run -it --rm -v <bids_root>:/bids_dataset bids-smriprep:latest python /run.py /bids_dataset --participant_label 01 02 03 -MNInormalization -hsvs_5ttgen -cleanup
/bids_dataset: 容器内输入BIDS路径,通过本地路径挂载(-v);participant_label: 指定分析某个或者几个被试,后面跟需要处理的被试,否则默认按照顺序分析所有被试;cleanup: 删除临时目录;MNInormalization: 将个体空间图像配准到标准空间的操作;hsvs_5ttgen: 将T1图像划分为5类组织,该选项需要完成freesurfer预处理;- 必须按照
sub-[num]_ses-[num]_[模态]格式命名,否则会报错。
3. dMRIPrep批处理dMRI影像
docker run -it --rm --gpus all -v <bids_root>:/bids_dataset bids-dmriprep:latest python /run.py /bids_dataset /bids_dataset/derivatives/dmri_prep participant --participant_label 01 02 03 -mode complete
participant_label: 指定分析某个或者几个被试,后面跟需要处理的被试,否则默认按照顺序分析所有被试;mode complete: 进行完整分析。- docker中的run.py要求必须有dwi.json文件。
4.dMRIrecon批处理
默认运行:
docker run -it --rm -v /bids_root:/bids_dataset bids-dmrirecon:latest python /run.py /bids_dataset /bids_dataset/derivatives/dmri_recon participant -mode connectome -bundle_json /scripts/bundle_list_all72.json -wholebrain_fiber 1000000 -fiber_num 2000 -atlases AAL1PD25_MNI desikan_T1w hcpmmp_T1w schaefer100x7_MNI schaefer100x7_T1w schaefer100x17_MNI schaefer100x17_T1w schaefer200x7_MNI schaefer200x7_T1w schaefer200x17_MNI schaefer200x17_T1w schaefer400x7_MNI schaefer400x7_T1w schaefer400x17_MNI schaefer400x17_T1w -cleanup
mode: 对应不同的分析模式,共有tract,dti_para:对应生成DTI参数;alps:对应生成TDI;dki_para:对应生成DKI参数;noddi_para:对应生成NODDI参数;connectome:对应生成脑结构网络几种,其中默认为tract,对应白质纤维束自动分割;/bids_dataset: 容器内输入BIDS路径,通过本地路径挂载(-v);/bids_dataset/derivatives/dmri_prep: 输出路径;participant: 个体被试水平顺序执行;bundle_json [str]: 需要追踪的纤维束名字的json文件(完整纤维束路径/scripts/bundle_list_all72.json)。wholebrain_fiber_num [int]: 全脑纤维追踪的streamline数量,默认10000000;atlases [str]: 指定分析图谱。对于后缀为T1w的图谱,需要预先对被试进行FreeSurfer分割!目前预定义的图谱见atlas_config_docker.json;- docker中的run.py同样要求
dwi.json文件; cleanup: 删除临时目录。
5.输出结果
DTI_mapping: 包含DTI的参数图,即FA/MD/RD/AD/DEC-map,以及在bundle mask上/纤维束tck上/脑区上的纤维束DTI参数统计信息json。其中DEC-map表示directionally-encoded colour map;fiber_tracts: 包含2个文件夹:fiber_tracts/bundle_segmentations,包含每个纤维束的bundle mask;fiber_tracts/Fibers包含纤维束tck文件,fiber_streamline.json是每个纤维束的统计信息;connectome: 包含Lookuptable和每一个脑图谱对应的结构连接网络。对于每一个脑图谱,*_connectome.csv表示脑连接定义为纤维束数量;*_meanlength.csv表示脑连接定义为纤维束平均长度;*_invnodevol.csv表示脑连接定义为纤维束数量+脑区体积矫正;*_dwispace.nii.gz是DWI空间的Label图;visualization: 可视化文件。其中Fibers_vtp包括每个纤维束;Fibers_bundlemask_vtp包括每个纤维束的整体mask;Fibers_endingmask_vtp包括每个纤维束两端mask。